Bacterias fluorescentes
Trabajando con la proteína verde fluorescente

Presentación
Aunque existen bacterias con fluorescencia natural, como la bacteria Pseudomonas fluorescens, con la que vamos a trabajar nosotros es Escherichia coli. Es más sencillo trabajar con ella, más seguro y además, los contenidos con los que vamos a trabajar solo tienen sentido si esa fluorescencia no es propia.
Lo que haremos entonces será transformar una cepa de la misma con el plásmido pGLO, para obtener la Proteína Verde Fluorescente. Esto es muy sencillo de realizar con el kit de Biorad y no necesita los estrictos requerimientos de los protocolos habituales, ni siquiera esterilizar el medio de cultivo. Además es bastante económico. Una vez que tengamos nuestra cepa transformada, la podemos ir sub-cultivando durante años sin necesidad de realizar otra transformación.
Con esta cepa, podemos trabajar diversos contenidos del área de la Biología molecular de una forma motivadora y entretenida, sin necesidad de grandes recursos.
La proteína verde fluorescente
La proteína verde fluorescente (GFP, en inglés, green fluorescent protein) es una proteína producida por la medusa Aequorea victoria, que emite fluorescencia en la zona verde del espectro visible. El gen de esta proteína se utiliza como marcador en biología molecular.
La GFP es una proteína monomérica de unos 230 aminoácidos que forma una estructura terciaria conocida como barril beta, común en muchas otras proteínas fluorescentes caracterizadas posteriormente.
En esta proteína, el barril beta está formado por 11 cadenas e incluye una hélice alfa central que atraviesa el barril en toda su longitud. En esta hélice hay tres aminoácidos consecutivos que forman un cromóforo natural, de forma que cuando la GFP es iluminada con luz ultravioleta, produce una brillante fluorescencia verde.

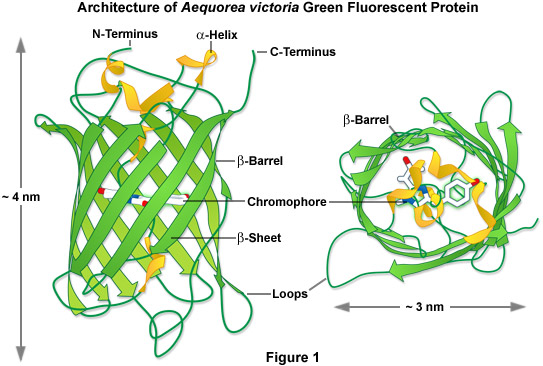
Antes de trabajar en el laboratorio, puedes visitar, el Protein Data Bank, PDB, y ver en tres dimensiones la proteína, conocer la secuencia de aminoácidos, estudiar su estructura secundaria y terciaria, encontrar los aminoácidos responsables de la florescencia, etc.
Hay muchas estructuras almacenadas, pero la principal es esta 1EMA
Esta proteína está codificada por en gen GFP, que está incluido en el plásmido pGLO.
El plásmido pGLO
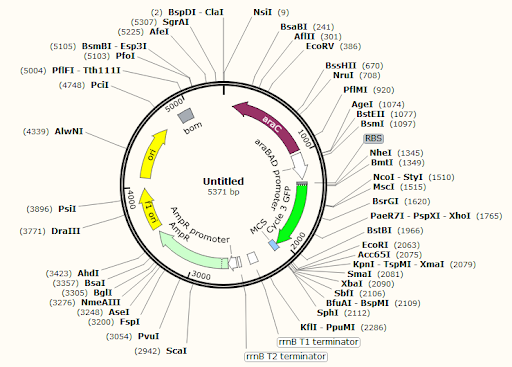
pGLO es un plásmido usado en biotecnología como vector, mide alrededor de 5500 pares de bases. Como todo plasmido, pGLO tiene una estructura circular en la que se expresan diversos genes. Entre ellos encontramos la β-Lactamasa, que proporciona resistencia al antibiótico ampicilina. También aparece el gen araC. Sin embargo, el gen más importante, y por el cual pGLO se usa como marcador, es el gen de GFP: Green Fluorescence Protein. GFP proporciona al ser transgénico fluorescencia al exponerlo a luz ultravioleta.
Podemos obtener la secuencia de ADN del plasmido y trabajar con el alumnado utilizando herramientas como SnapGene viewer, para profundizar un poco más en los genes que lo componen y su mecanismo de regulación. La imagen de la derecha es el resultado de pegar directamente la secuencia de plásmido en SnapGene. El software reconoce los genes implicados y nos los muestra.
Prácticas de laboratorio
Si no tenemos la bacteria fluorescente, la primera de ellas será, claro, la transformación. Como ya hemos comentado, es más sencilla de lo que parece y no necesitas un equipamiento especial. Una vez que tengamos nuestra cepa transformada, el simple hecho se sembrarlas en diferentes medios (una práctica corta y sencilla) me permite trabajar diversos contenidos como la resistencia a antibióticos, la regulación génica, etc.
